

CAVITY是一个基于结构的结合位点探测程序。对于包括分子对接、全新设计等许多应用来说,确定配体体结合位点都是非常关键的问题。CAVITY使用一种几何学的方法来寻找潜在的结合位点,并通过几何形状状以及物化信息,对结合位点进行判定。CAVITY可以预测结合位点的最大结合能,更为关键的是,CAVITY能够精确并清晰的定义结合位点的范围。
请注意:
1.LigBuilder v2.0采用刚性受体假设。也就是说,对于给定蛋白,您只需要运行一次CAVITY。
2. CAVITY运行很快。对于大多数蛋白来说,在目前的主流平台上,CAVITY需要运行几分钟时间。如果蛋白结构很大,运行时间会相应延长。但是CAVITY运行时需要占用大量内存,通常为1~2G。.
CAVITY运行方法:
cavity Parameter_file
例如:
./cavity 1db4.input
在执行前您需要仔细编辑该参数文件。文件中的关键词请参看后续章节。
为了方便用户操作,我们为一些不同的任务提供了一些默认的参数集,用户可以简单的通过关键词"INCLUDE"调用这些参数集。其路径为 "LigBuilderV2/default/", 包括5个参数集:
Overall default set(全局参数):
cavitydefault.input: 默认的全局参数,包括所有默认参数
Detection mode set(探测模式参数集):
standard.input: 用于标准探测模式,针对普通的结合位点,是当前默认参数集。
peptide.input: 针对较浅的结合位点的参数集,例如对多肽结合位点、蛋白-蛋白界面
large.input: 针对较大的结合位点的参数集,例如对大的蛋白蛋白界面,多功能位点,通道,核酸结合位点等
super.input: 针对特别大的结合位点的参数集,例如对大的通道、大的多聚界面
Ligand-locate mode set(配体定位模式):
locate.input: 该参数集启动配体定位模式,在该模式下,CAVITY忽略离已知配体较远的蛋白单体 (依赖于LIGAND_FILE)
DETECT_MODE: CAVITY探测模式。 0: 全蛋白模式,该模式下 CAVITY探测整个蛋白并寻找所有潜在结合位点,该模式为默认模式。1: 配体模式,该模式下CAVITY仅探测给定Mol2文件附近的结构。这帮助程序寻找真正的结合位点。大多数情况下,CAVITY可以在未知配体的情况下找到结合位点,如果你对全蛋白模式的探测结果不满意,也可以采用配体模式。2: 区域模式, 该模式下CAVITY仅探测用户指定的空间范围。用户可以通过该模式精确指定结合位点,使得LigBuilder v2.0不对用户不想要的区域进行设计。
RECEPTOR_FILE: PDB 格式的受体文件。CAVITY运行的必须文件。由于诱导契合效用,我们推荐使用复合物的结构而不是非结合态的结构。
LIGAND_FILE: Mol2 格式的配体文件(仅在DETECT_MODE 为1时必须).该文件指引CAVITY程序的探测位置,如果该配体的大小、形状以及位置合适,探测结果会更为完美。使用从复合物中提取出的结构即可完成该任务。
PARAMETER_DIRECTORY: "parameter"目录的路径。该目录下包括LigBuilder v2.0所使用的力场参数。原子、键以及二面角参数来自于Tripos力场。
MIN/MAX_X/Y/Z: 区域探测盒子的6个顶点坐标。( 在 DETECT_MODE是2时必须)
HETMETAL: YES: 考虑蛋白中的金属离子, NO: 与YES相反.,默认: YES
HETWATER: YES: 考虑蛋白中的水分子, NO: 与YES相反.,默认: NO
OUTPUT_RANK: CavityScore的限制。 CAVITY不会输出CavityScore低于该值的探测结果。用户可以通过提高该数值避免CAVITY输出无意义的结果。
JUDGE: 探测算法: 0:界面模式。根据界面形状探测,该模式在蛋白蛋白界面探测上有优势。 1:体积模式。根据空间形状进行探测,该模式在大型结合位点探测上有优势。 2:界面-体积模式。该模式适合探测较为紧凑的结合位点,并且能够较为清晰的判定结合位点边界。对药物设计来说,第三种模式往往更为合适。对单纯的结合位点探测来说,第二种模式更具备对多种蛋白的适应性。默认: 2.
RADIUS_LENTH: 擦除球的半径(单位: 0.5 ang.). 探测很平坦或者狭窄的结合位点时可适当增加该值。默认: 10
SEPARATE_MIN_DEPTH: 默认结合位点最小深度。 深度不足该值的区域连接区会被切分。
SEPARATE_MAX_LIMIT: 默认界面面积。如果实际结合位点大于探测结果,可以适度增加该值,反之亦然(仅JUDGE为0时有效)
MAX_ABSTRACT_LIMIT: 默认核心界面面积。如果实际结合位点大于探测结果,可以适度增加该值,反之亦然。(仅JUDGE为0时有效)
MAX_ABSTRACT_DEPTH: 默认核心深度。如果实际结合位点大于探测结果,可以适度增加该值,反之亦然(仅JUDGE为1 或 2时有效)
MAX_DEPTH_VACANT: 默认深度。(仅JUDGE为1 或 2时有效)
MAX_ABSTRACT_LIMIT_V: 默认核心体积。如果实际结合位点大于探测结果,可以适度增加该值,反之亦然。(仅JUDGE为2时有效)
SEPARATE_MAX_LIMIT_V: 默认体积。 如果实际结合位点大于探测结果,可以适度增加该值,反之亦然。(仅JUDGE为2时有效)
输出文件将会加上RECEPTOR_FILE 的前缀,并后缀以结合位点编号。
下述的两个输出文件存储了对于BUILD模块最为重要的信息。请避免手动编辑这两个文件。
name_pocket.txt: 结合位点区域的原子列表。文本格式。 (BUILD模块所需)
name_grid.txt: 结合位点区域的格点列表。文本格式。(BUILD模块所需)
CAVITY输出如下可视格式的文件,用以帮助用户检查探测结果。
name_surface.pdb: 此文件包含结合位点的形状信息以及打分信息。该文件为PDB格式,用户可以使用分子建模软件打开该文件来观察结合位点的几何形状。用户也可以使用文件编辑器查看该文件,以查看Cavity对该结合位点结合强度的预测。如果预测结合强度小于6.0(Kd相当于1uM)时,说明该结合位点可能不是一个合适的药物设计位点。
name_vacant.pdb: 此文件包含结合位点的体积信息以及打分信息。该文件为PDB格式,用户可以使用分子建模软件打开该文件来观察结合位点的结合形状。
name_cavity.pdb:此文件包含结合位点的原子信息。该文件为PDB格式,用户可以使用分子建模软件打开该文件来观察结合位点所涉及的原子和残基。它也是"name_pocket.txt"的可视化形式。
注意:有些分子建模软件可能无法正确显示这些文件,如果显示失败,请尝试使用不同的软件。(推荐使用Pymol来查看这些文件)
CAVITY会判断结合位点中的关键相互作用位点,以此为基础生成药效团模型。涉及到下列文件:
name_key_site.pdb: 该输出文件包含关键相互作用位点信息,为PDB格式,其中蓝色的氮原子表示氢键给体位点,红色的氧原子表示氢键受体位点,白色的碳原子表示疏水位点。
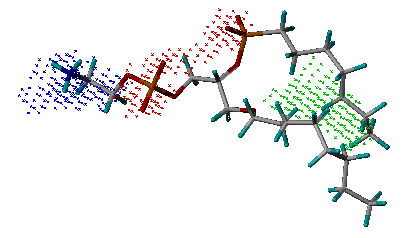
关键作用位点示例
(Phospholipase A2, PDB entry 1POE)
name_pharmacophore.pdb: 该输出文件为药效团模型,为PDB格式。颜色定义方法与"name_key_site.pdb"中相同。
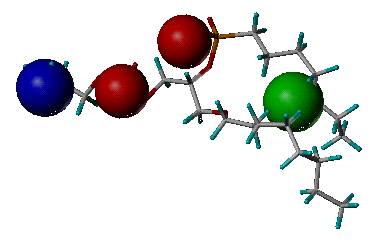
药效团模型示例
(Phospholipase A2, PDB entry 1POE)
name_pharmacophore.txt: 该输出文件包含药效团模型的信息。该文件列出可所有药效团的强度以及相互距离,并根据强度排序。该文件是"name_pharmacophore.pdb"的补充。
[Content] [Introduction] [Download] [Install] [Overview] [CAVITY] [BUILD] [Skills] [FAQs]
(These web pages are edited by Yaxia Yuan. Latest update: Jan., 2012)