

BUILD模块的主要功能是通过基于片段的策略构建针对目标蛋白的配体, 设计过程主要采用了遗传算法。该模块通过内置的合成分析系统,对设计的分子的可合成性进行控制。此外,通过一系列优化规则,使设计的分子合理、高效的匹配目标受体。同时,运用一系列的ADME/T和类药规则,并在设计过程中使用类药片段,以提高设计结果的制药成功率。除这些新的特性之外,BUILD继承LigBuilder v1.2中所有的功能,并提升了GROW和LINK模块的性能。BUILD会将所有的设计结果编织为一个报告文件。
请注意:
1.BUILD依赖于CAVITY模块所生成的结合位点描述信息。因此,使用BUILD前需要先用CAVITY模块进行结合位点分析。
2.虽然BUILD本身可以作为单线程程序运行,但单线程运行会需要非常长的时间来完成一个设计任务。 通常来说,用户应当使用多个CPU来加速设计过程。加速效应与使用的CPU数近似为线性关系。BUILD模块提供Automatic模式来帮助用户完成并行化过程。运行于主流的工作站时,一个典型的计算任务消耗1~5千CPU小时。如果结合位点的可药性弱,运行时间会相应变长。
3.BUILD会使用大量的内存,一个典型的BUILD进程会使用1~2G内存,用户也可以通过调用不同的内存控制参数集来调整内存使用策略。
BUILD运行方式:
build -Function Parameter_file [Id]
例如:
./build -Automatic build.input
在执行前您需要仔细编辑该参数文件。文件中的关键词请参看后续章节。
为了方便用户操作,我们为一些不同的任务提供了一些默认的参数集,用户可以简单的通过关键词"INCLUDE"调用这些参数集。其路径为 "LigBuilderV2/default/", 包括如下参数集:
Overall default set(全局参数):
default.input: BUILD的所有默认参数。
Default path(路径):
path.input: 所有输入输出文件的默认路径以及路径模板。因为BUILD模块的Automatic模式需要根据一定的命名规则进行后续处理,用户尽量不要直接修改这些路径,而是通过定义任务名前缀的方式调整输入输出的位置。
Diversity set(多样性):
diversity_low/moderate/high.input: 三种默认的多样性控制策略。增加多样性会降低可合成性以及计算速度。默认:low。
Synthesize set(合成):
synthesize_knowledge/rapid/moderate/accurate.input: 四种合成策略,用于调整计算速度与精度间的平衡。如果对计算精度不进行限制,内置的合成分析器会消耗极多的资源。根据我们的经验,使用略为低精度的可合成性分析策略效率较高精度的策略更好。虽然低精度的策略会降低分析的可靠性,但更高的计算速度使得设计结果大量增加,可以减小低可靠性的影响。基于knowledge的方法在计算资源受限时非常有用,默认情况使用rapid策略。
Optimization set(优化):
optimize_none/soft/moderate/intense.input: 四种优化策略,调整分子在力场中的优化幅度。默认: Intense(Exploring模式), soft(Growing/Linking模式)。
Memory control set(内存控制):
design-memory_500M/1G/2G.input: 设计所占用的内存。增加内存可以加速遗传算法效率。默认: 1G/每进程
synthesize-memory_600M/1G/2G.input: 内置合成分析器所占用的内存。增加内存可以加快合成分析速度。默认:1G/每进程
analysis-memory_1G/2G/3G.input: 后置合成分析器所占用的内存。增加内存可以加快合成分析速度。默认: 1G/每进程
注意:每个进程所占据的最大内存为第一项与第二项的和(设计阶段),或者是第三项(后处理阶段)。用户需根据自己的系统资源选择合适的内存策略。
Recommend set(结果挑选):
recommend_redock/inclusiveness/moderate/exactness.input: LigBuilder v2.0结果挑选器的四种默认策略。对每个设计任务,BUILD模块通常会输出非常多的设计结果(100万~1000万个分子),挑选器可以帮助用户挑选出最有潜力的分子。挑选器使用基于大量设计结果统计的动态评估算法来确定挑选阈值,因此通常情况下,这种方法必须在分子集较大时才能发挥作用。四种策略的过滤强度依次增加,在使用'exactness'策略失败时,可以尝试'moderate'或'inclusiveness'策略。如果用户想使用挑选器来精选dock结果,可以使用'redock'策略。默认: exactness(Exploring模式), inclusiveness(Growing/Linking模式)。
Special design strategy set(特殊设计策略):
lead.input: 先导设计模式。设计低分子量,高原子经济性的先导结构。
mimic.input: 仿制模式。基于已知抑制剂设计相似的分子。(Growing/Linking Mode)
Speedup strategy set(加速策略):
speedup.input: 如果用户的计算资源非常有限或者希望尽快完成一次设计工作,强烈推荐使用该加速策略。该策略限定仅探索较小的化学空间,因而效率较高。并且,该策略与正常计算相比,能够获得近似的设计质量,仅仅是分子多样性较低。
Usersettings(策略设定):
usersettings.input: 用户可以通过该文件调用设计时所使用的策略。
DESIGN_MODE: LigBuilder v2.0的核心功能是设计匹配目标蛋白的分子。LigBuilder v2.0支持三种设计策略,也即是1: Growing strategy(生长模式), 2: Linking strategy(连接模式)以及 0: Exploring strategy(探索模式)。运用生长模式时,用户需要提供已经预先放置入结合位点的"seed(种子)"结构 ,然后LigBuilder v2.0以此结构为起点设计分子。 当用户已经有合适的先导化合物,想要在提升其活性时,可以采用该模式(先导优化)。使用连接模式也需要用户提供种子结构,且必须提供超过1个的种子。同生长模式相同,种子需要预先放置入结合位点,并且最好能与蛋白形成一定的相互作用。然后LigBuilder会尝试将这些种子整合成一个完整的分子。该策略可以将多个关键片段融合成一个分子(片段连接)。此外,LigBuilder v2.0版提供了从已知抑制剂中提取种子结构的功能,并且能够基于这些片段设计新的分子。使用这种方法,能够高效的设计仿制药物。运用探索模式时,不再需要用户准备种子结构。LigBuilder会自动生成种子结构并在设计过程中不断更新这些结构。该策略具有强大先导化合物发现与从头设计能力。
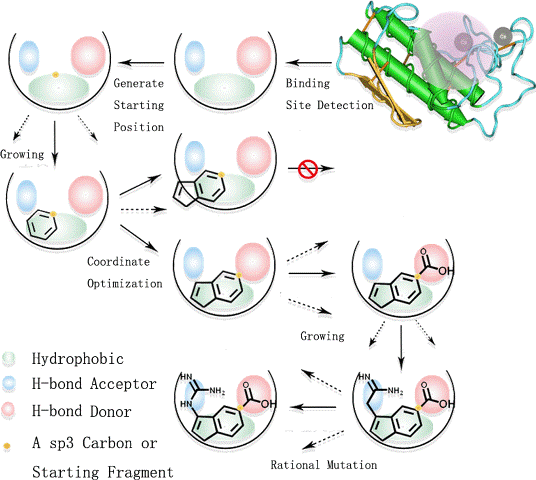
设计过程示意图
POCKET_ATOM_FILE: 由CAVITY提供的原子文件(必要文件)。
POCKET_GRID_FILE: 由CAVITY提供的格点文件(必要文件)。
SEED_LIGAND_LIST:Mol2格式的"seed"结构列表 (DESIGN_MODE:1 or 2)。如上所述,对生长模式和连接模式来说这是必要文件,因为这是所有设计结果的母体。因此,该种子结构最好能够与结合位点有较强或者关键的相互作用。LigBuilder v2.0可以通过"Extract(提取)"功能从已知的抑制剂拆分种子结构,这里要求这些抑制剂已经被正确的对接到受体结合位点或者是来自于晶体结构。关于种子结构准备工作更多的细节,请参阅 高级技巧 章节 。
ADD_HSPC: YES: 视种子结构中的所有氢原子为生长位点。 NO: 仅使用用户定义的生长位点。 (DESIGN_MODE:1 or 2)
接下来,这些参数用于自动化模式启动伪并行计算:
CONTINUE_MODE: YES :恢复运算时继承种子库以及输出文件。No : 恢复运算时覆盖输出文件,并重新生成种子
SESSION_NUMBER: 一个设计任务运行的总进程数。 通常来说,至少为50,默认为100,越多越好。如果用户的计算资源有限,请运用前面所说的加速策略,否则计算时间可能会非常长。
MOLECULE_NUMBER: 一个任务的目标分子数。 通常来说,至少为10万,默认100万。 如果用户的计算资源非常充足,我们推荐一个任务设计1000万个分子。 (参考计算速度: 1千分子/ CPU 小时)
OPTIMIZE_SEED: YES: 使用LigBuilder v2.0中的力场参数优化种子结构 NO: 保持原有坐标。如果种子结构放置不佳,可能会导致碰撞。(DESIGN_MODE:1 or 2):
LIGAND_COLLECTION_FILE : 此为BUILD模块的输出文件。整个设计过程中,所有符合标准的分子全部被输出到该文件中。文件格式为LigBuilder自己的LIG格式,可以被“Process”功能转换为Mol2格式。
LEAD_COLLECTION_FILE : 此为BUILD模块生成的种子记录,Mol2格式。
SEED_RECORD_FILE : 当前种子库,Mol2格式。
RANDOM_SEED : 随机数生成器的种子。 Natural number(自然数):人工指定该数为种子。 -1: 根据系统时间取种子(默认). -2: 根据任务ID取种子(需要任务Id). 根据系统时间取种子,则不同进程的计算结果完全不同,并且每个进程的计算过程都不可重现,哪怕是系统环境以及启动时间都完全相同。根据ID取种子时,环境不变时ID相同计算结果相同,具有重现性。人工指定种子,指定相同的数也具有重现性。
输出规则包括"APPLY_CHEMICAL_RULES",
"APPLY_FORBIDDEN_STRUCTURE_CHECK",
"APPLY_TOXIC_STRUCTURE_CHECK",
"MAXIMAL_MOLECULAR_WEIGHT",
"MINIMAL_MOLECULAR_WEIGHT",
"MAXIMAL_LOGP", "MINIMAL_LOGP", "MAXIMAL_HB_DONOR_ATOM", "MINIMAL_HB_DONOR_ATOM", "MAXIMAL_HB_ACCEPTOR_ATOM", "MINIMAL_HB_ACCEPTOR_ATOM", "MAXIMAL_PKD", "MINIMAL_PKD" and "MINIMAL_AVER_PKD"。请参考高级技巧章节。
MAXIMAL_RESULTS: 每个独立进程的设计目标数。 0: 不限制 Natural number: 当收集到足够分之后终止。
BUILD使用遗传算法来进化分子。大略的运行方式如下:(1)基于种子结构生成初始种群;(2)从种群中选择母本进入交配池;(3)将种群中的部分精英直接送入下一代种群;(4)通过交配池衍生出足够的分子填满下代种群;(5)重复2~5的过程,直到达到限制。
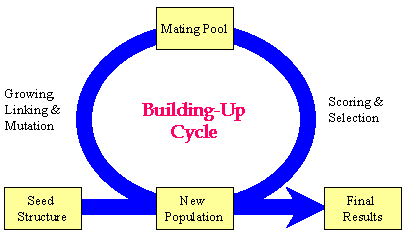
BUILD中遗传算法示意图
如下参数为遗传算法的控制参数:
NUMBER_OF_GENERATION: 遗传代数(遗传算法循环数)。通常8~15代就足够了。
NUMBER_OF_POPULATION: 种群大小。该参数会影响程序使用的内存大小。通常是几千。
NUMBER_OF_PARENTS: 选入交配池的母本数量。推荐NUMBER_OF_POPULATION / NUMBER_OF_PARENTS 的比值大于10。
SIMILARITY_CUTOFF: 交配池中的相似度阈值。如果该值为1,则允许相同的分子同时进入交配池。该值越低,整体的多样性越高。推荐该参数为0.8~1.0。
ELITISM_RATIO: 精英分子的百分比。该参数取值为0到1之间。例如,如果设为0.10,也就是说当前种群中排名前10%的分子都会直接进入下一代种群。这样可以保证精英分子不会在计算过程中遗失。推荐该数值在0.1附近。
GROWING_PROBABILITY: 对当前版本的BUILD来说, 生长操作是必须的,因此该值必须为1.00。
LINKING_PROBABILITY: 对当前版本的BUILD来说, 连接操作是必须的,因此该值必须为1.00。
BUILDING_BLOCK_LIBRARY: 基础分子构建单元的库。默认为 "fragment.mdb".
BUILDING_BLOCK_LIBRARY_EXTEND: 扩展分子构建单元库,默认为"fragment.mdb/extend".
BUILDING_BLOCK_LIBRARY_ROTATABLE: 多构象构建单元库,默认为 "fragment.mdb/rotatable".
FORBIDDEN_STRUCTURE_LIBRARY: 禁用子结构的库,默认为 "forbidden.mdb".
TOXIC_STRUCTURE_LIBRARY: 毒性子结构的库,默认为"toxicity.mdb".
SYNTHESIZE_LIBRARY: 合成库,默认为"synthesize.mdb".
如果用户是初学使用LigBuilder v2.0,可以直接使用这些默认设置。关于自定义库可以参考高级技巧章节。
输出目录会加上受体名的后缀,主要包括如下几个文件:
result/output_name/ligand.lig: 包含BUILD模块设计的所有结果,LigBuilder的LIG格式。
result/process_name/INDEX: 通过"Process"功能获得的分子列表,由LIG文件转换来。
result/cluster_name/INDEX: 通过"Cluster"功能得到的聚类列表。
result/synthesize_name/synthesize.log 通过"Synthesize"功能得到的合成分析信息。
result/report_name/report.html: HTML格式的最终结果报告。
[Content] [Introduction] [Download] [Install] [Overview] [CAVITY] [BUILD] [Skills] [FAQs]
(These web pages are edited by Yaxia Yuan. Latest update: Jan. 2012)